清华大学信息国家研究中心数基生命系统交叉创新群体成员、自动化系汪小我教授与美国斯坦福大学统计系王永雄教授团队合作,开发了神经网络解释算法NeuronMotif,实现了从神经元中自动归纳和提取基因调控序列编码规则。研究成果以“NeuronMotif: 通过深度神经网络的逐层解耦破译基因顺式调控编码(NeuronMotif: Deciphering cis-regulatory codes by layer-wise demixing of deep neural networks)”为题,于2023年4月6日发表于《美国科学院院刊》(PNAS)期刊上。论文链接:https://www.pnas.org/doi/10.1073/pnas.2216698120。
近年来,大数据驱动的深度神经网络模型已在多个领域取得了重要进展。例如,在蛋白质结构和基因调控功能预测等方面实现了高精度的预测。然而,这些模型通常是不透明的黑盒,难以理解。透明化神经网络黑盒可以帮助我们获取模型从数据中学习到的知识,从“只知其然”转变为“知其所以然”,对于促进神经网络理论发展和增进相关领域的认知具有重要意义。例如,基因调控序列是基因组上能够调控基因表达的DNA序列,决定了细胞的功能和状态;当前,卷积神经网络(CNN)等深度神经网络模型在基因调控序列的功能预测问题上能够取得很好的预测效果,但由于神经网络解释方法的缺失,使得研究者难以从这些深度模型中发现基因调控序列的深层编码规则,严重制约了基因调控规律解析和重构的研究进程。
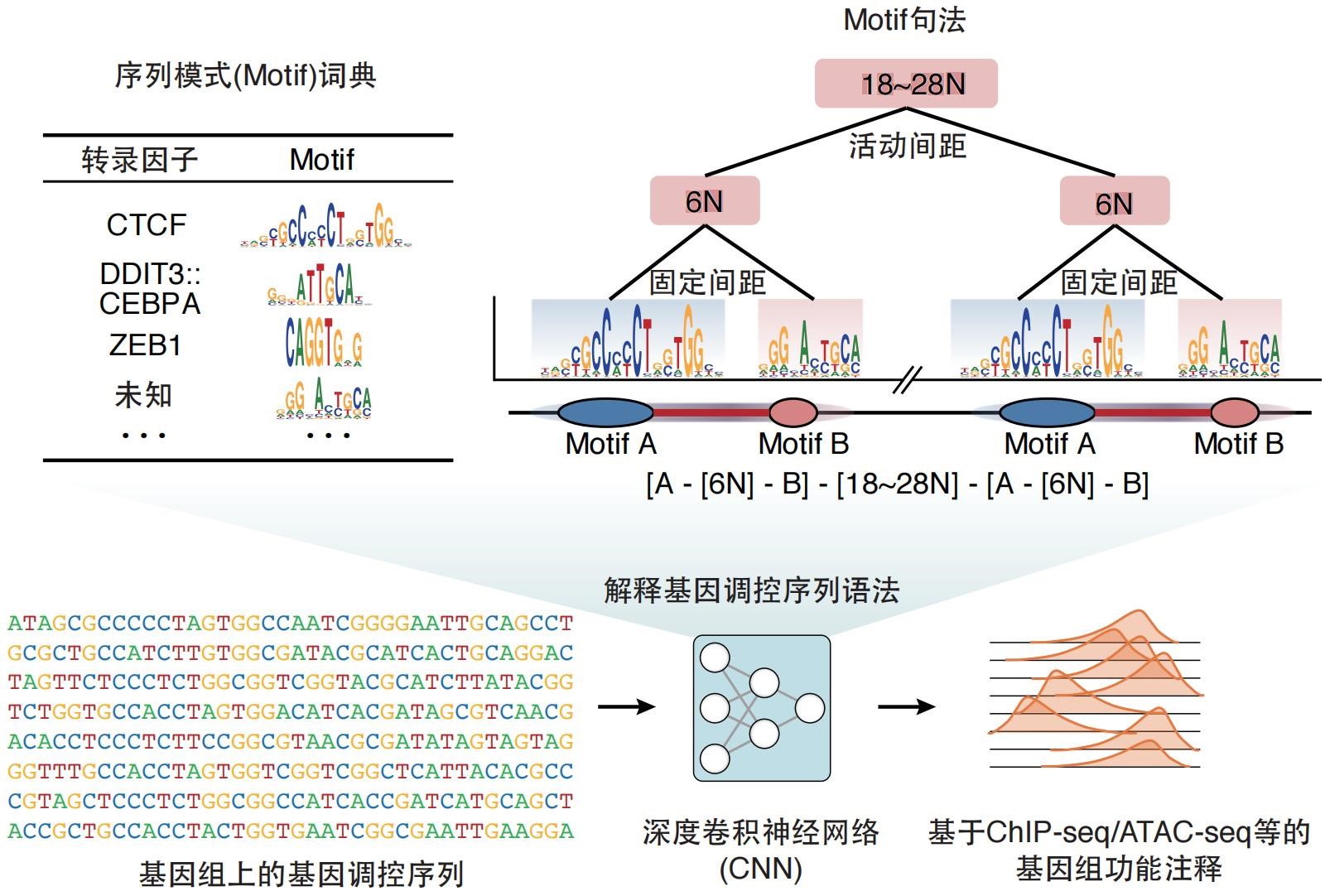
图:从深度卷积神经网络中提取基因调控序列的语法规则
研究团队发现,CNN解释困难的一个主要原因在于深层神经元大多都是“多面神经元”(multifaceted neuron),这种神经元能够同时被多种不同序列模式激活,直接对这些混合模式进行可视化往往只能获得难以被人理解的结果,而CNN中的最大池化结构是导致深层神经元识别多种序列模式的关键原因。针对多面神经元问题及其形成机制,研究团队提出了NeuronMotif方法,该方法首先基于蒙特卡洛采样和遗传算法得到大量能充分激活神经元的序列集合,然后计算这些序列在神经网络各层的特征图,并通过反向逐层聚类的方式分离不同序列模式所对应的特征图,将具有不同模式的序列划分到不同的子集中,最终通过对每个子集的可视化获得易于理解的序列模式特征。在此基础上,该方法构建了基于结构化语法树的自动化知识提取方法,从数据中自动归纳转录因子结合位点序列模式、组合模式、间距、次序等调控序列语法规则。
NeuronMotif归纳提取的调控序列语法规则能够得到文献和多种生物组学数据的支撑和验证。通过对人类基因组数据的学习,NeuronMotif发现了数百种序列模式,与数据库中的已知基因调控序列模式高度匹配;通过NeuronMotif获取的深层神经元语法规则与多种细胞/组织中的染色质开放区域模式高度匹配,并得到了基因表达数据的支持;序列模式的组合和排布等规律在跨物种基因组序列上具有显著的序列保守性。
NeuronMotif为我们提供了一种解读深度神经网络模型中深层神经元识别模式的新方法。其解释结果还可用于人工神经网络的诊断和改进,有助于降低神经网络调参的困难。借助该方法,我们可以通过神经网络的训练和解释,从数据中获取可供人类专家理解的知识,帮助我们更加深入地理解胚胎发育、疾病发生等生物过程中的基因调控规律,并为基因治疗等应用中定制化逆向构造人工基因调控序列提供支撑。
清华大学自动化系博士后魏征为本文的第一作者,清华大学汪小我教授和美国斯坦福大学王永雄教授为本文的共同通讯作者。清华大学李衍达教授、张学工教授、江瑞教授、魏磊助理研究员、花奎博士,斯坦福大学博士后马士宁也对本文做出了重要贡献。该研究得到了国家自然科学基金、国家重点研发计划、北京信息科学与技术国家研究中心交叉创新群体、清华大学国强研究院、北京智源人工智能研究院等的资助。

